资料来源:CC0 Public Domain
通过识别导致癌症的基因突变,科学家们在理解和开发许多癌症的治疗方法方面取得了重大进展。现在,由威尔康奈尔医学院、纽约长老会医院和纽约基因组中心(NYGC)的研究人员领导的一个团队开发了一种机器学习技术,用于检测DNA的其他具有类似效果的修改。
这项研究发表在5月10日的《纽约时报》上癌症的发现《美国癌症研究协会》(American Association for Cancer Research)的一份杂志关注的是一种名为甲基化的DNA化学修饰,这种修饰通常会使附近的基因沉默。这项新技术可以分析检测到的数千个DNA甲基化变化肿瘤细胞并推断出哪些基因可能推动肿瘤生长。
甲基化是一种“表观遗传”过程,通常通过改变DNA结构而不改变基因中包含的信息来调节整个基因组的基因活性。然而,偶尔,过度甲基化,称为超甲基化,发生在附近肿瘤抑制基因使基因沉默,并帮助触发或驱动失控的细胞分裂癌症。
“如果我们能用这样的技术分析大量的肿瘤,我们就可以绘制出导致肿瘤的表观遗传变化肿瘤的生长资深作者丹·兰道博士说,他是血液学和肿瘤医学部门的医学副教授,也是威尔康奈尔医学桑德拉和爱德华·梅耶癌症中心的成员。“然后我们可以利用这些信息来提高我们对癌症起源的理解,以及优化个别患者的治疗方法。”
这项新技术所面临的挑战类似于癌症研究人员在DNA突变方面所面临的挑战——如何区分“驱动”突变和更丰富的“乘客”突变,后者对癌症没有影响。尽管现在有复杂的方法来区分基因突变兰道博士说,在美国,区分司机甲基化变化和乘客甲基化变化的技术还远没有那么复杂。兰道博士也是NYGC的核心成员,也是纽约长老会/威尔康奈尔医学中心(NewYork-Presbyterian/Weill Cornell Medical Center)的肿瘤学家。
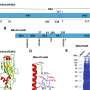
的新算法由兰道博士的团队开发的,叫做MethSig。它利用现有的信息,例如基因组特定区域的甲基化背景率,来估计给定的甲基化变化何时可能是癌症的驱动因素。
研究人员将该算法应用于不同肿瘤类型的DNA甲基化图,发现它可以推断出少量的癌症驱动事件——每个肿瘤中位数为12个左右——与数千个乘客甲基化变化相比。推断的驱动因素甲基化模式在患者和肿瘤类型之间是一致的,以及其他统计特征表明,与现有方法相比,该算法的性能无增量提高。
研究小组通过敲除慢性淋巴细胞白血病(CLL)细胞中受影响的基因,进一步验证了几个最强有力的DNA甲基化癌症驱动因素,并表明当细胞未经治疗时,该基因的缺失促进了细胞的生长,同时也存在一些标准的CLL治疗。总的来说,研究人员得出的结论是,他们的算法检测可能导致癌症的甲基化变化比目前的方法更加敏感和选择性。
为了证明该算法在改善癌症预后和治疗方面的潜力,研究人员将MethSig应用于另一组CLL样本,并使用其推断来预测个别患者癌症的侵袭性。
“我们使用MethSig开发的分类器为每个患者提供了估计的风险,我们发现估计风险较高的患者更有可能有更糟糕的结果,”第一作者Heng Pan博士说,他是威尔康奈尔医学院阿尔瓦利德王子·宾·塔拉勒·宾·阿卜杜勒阿齐兹·沙特计算生物医学研究所的高级研究助理,他执行了该研究中的大部分实验。
研究人员计划使用更多的癌症数据集和更全面的基因组信息继续使用和改进MethSig算法。
“最终,我们设想能够绘制出癌症驱动DNA甲基化变化的整个图景肿瘤因此,我们可以将精准医疗的范围扩大到遗传学之外,还包括癌症表观遗传变化的关键维度。”兰道博士说。
“信息论”被招募来帮助科学家寻找癌症基因
期刊信息:癌症的发现
所提供的威尔康奈尔医学院